고정 헤더 영역
상세 컨텐츠
본문
1. DNA 서열의 돌연변이
폴리펩티드를 코딩하는 서열을 코딩 영역 또는 오픈 리딩 프레임(ORF)이라고 합니다.
ORF 내의 각종 돌연변이는 폴리펩티드 산물의 아미노산 서열에 있는 변화를 일으키는 원인이 될 수 있거나, 일으키는 원인이 되지 않을 수 있습니다. DNA의 돌연변이가 폴리펩티드의 아미노산 변화를 일으키는 원인이 되는 경우, 그것은 미스센스 또는 비동의성 돌연변이로 불립니다. 돌연변이가 폴리펩티드의 아미노산 변화를 일으키지 않는 경우, 그것은 침묵 돌연변이 또는 동의어 돌연변이라고 불립니다. 종래의 통념에서는 아미노산에는 변화가 없기 때문에 동의 변이는 단백질의 기능을 변화시키지 않는 것으로 생각되고 있으나 최근 연구 결과는 많은 단백질에서 동의 돌연변이가 단백질의 입체 구조의 변화를 초래하기 때문에 단백질의 기능도 변할 수 있음을 보여줍니다. 단백질의 접힘은 번역과 동시에 진행되는 과정이기 때문에 적절한 단백질의 접힘은 번역 속도와 밀접한 관련이 있습니다. 코돈의 사용에 영향을 미치는 동의어 돌연변이는 이 과정을 혼란스럽게 하여 실수로 접힌 폴리펩티드를 초래할 수 있습니다. 사실, 일부 사람의 질병은 그러한 동의어의 돌연변이와 관련이 있을 수 있습니다.
2. RNA의 몇 가지 특징
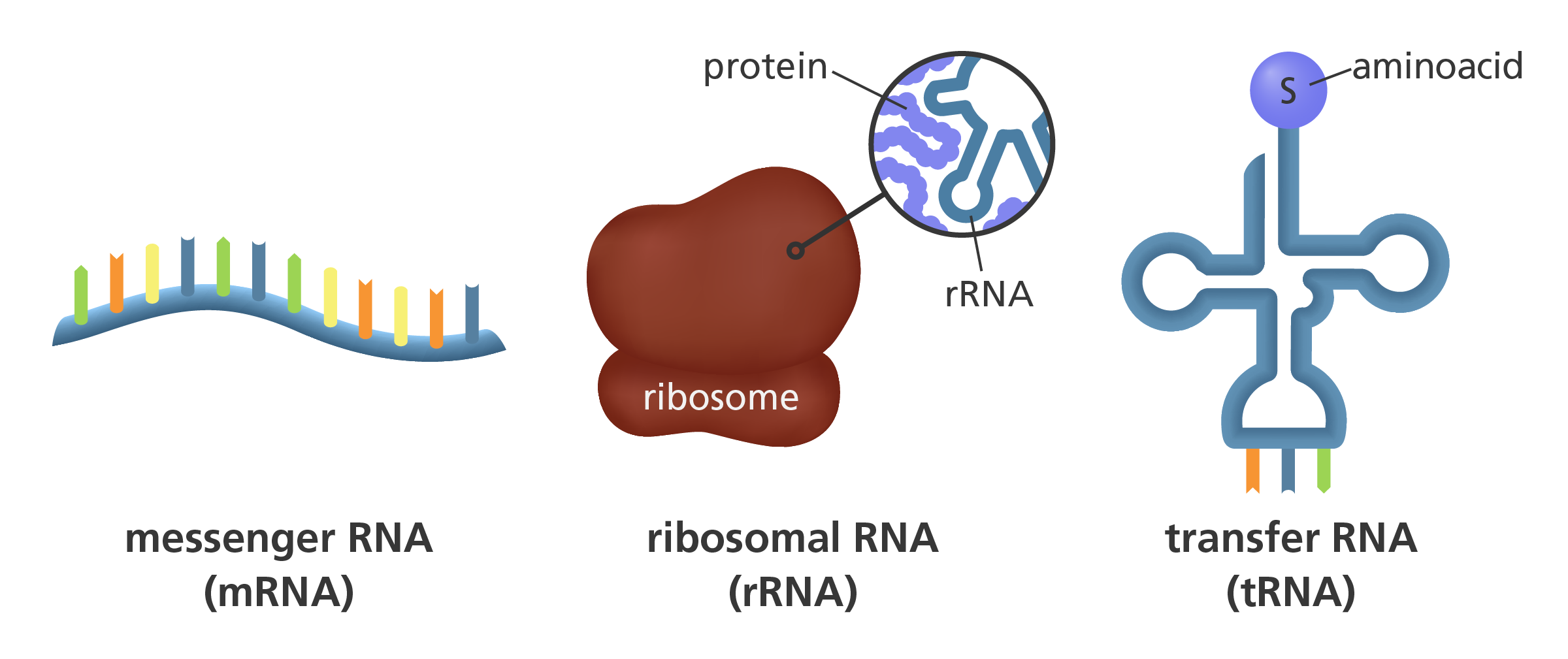
전통적인 분자 생물학에서 RNA에 대한 논의는 단백질 합성과 관련된 세 가지 RNA, 즉 리보솜 RNA (rRNA), 메신저 RNA (mRNA) 및 트랜스퍼 RNA (tRNA)에 중점을 두었습니다. 그중 rRNA와 tRNA는 비코딩이지만 mRNA는 비코드입니다. 단백질 코딩. 그 이후로 기능적인 비 코딩 RNA 분자의 세계는 크게 확장되었습니다. 위에서 언급했듯이 RNA는 레트로 바이러스 유전 물질입니다. RNA 분자는 염기 상보성에 의해 분자가 접혀 이중 가닥 세그먼트를 형성하는 영역을 제외한 단일 가닥입니다. DNA와 마찬가지로 RNA는 뉴클레오티드 (리보 뉴클레오타이드)로 구성됩니다. 그러나 DNA와는 두 가지 차이점이 있습니다. 당이 리보스인 것과 "T"대신에 염기 우라실("U")이 존재하는 것입니다. 따라서 염기쌍은 "A"와 "U" 사이에 있습니다. 번역과 관련된 세 가지 RNA (rRNA, mRNA, tRNA) 중에서 mRNA에 중점을 둡니다.
2.1 mRNA 불안정성
mRNA를 쉽게 분해하는 효소 RNAse가 편재하여 존재하는 것과는 별도로, mRNA의 구조 자체도 그 불안정성에 기여하고 있습니다. 리보스 당은 특히 알칼리성 pH에서 RNA를 DNA보다 불안정하게 만듭니다. 알칼리성 pH에서, 리보스 당의 2'-OH는 알칼리 가수분해를 겪고, 이에 따라 인접한 뉴클레오티드 사이의 인산 결합이 절단되어 2'-3' 원형 뉴클레오티드가 형성됩니다. 이 뉴클레오티드는 리보뉴클레오사이드 2' 및 3' 단일 포스페이트 생성물의 혼합물을 생성합니다. 대조적으로, DNA는 20개의 탄소에 OH 대신에 H가 있어, 2'-3'의 원형 뉴클레오티드의 형성을 방해합니다. 이렇게 하면 알칼리성 가수 분해가 방지되고 DNA는 알칼리성 pH에서 안정화됩니다. 그러나 산성 pH에서는 DNA와 RNA 모두에서 포스포디에스테르 결합의 가수분해가 일어납니다. RNA는 특히 37℃ 부근에서 급속한 알칼리 가수분해를 받기 때문에 NaOH를 사용합니다.
RNA를 변성시키는 것은 권장되지 않습니다.
2.2 mRNA의 5' 및 3' 비 번역 영역
전형적인 진핵생물의 mRNA에는 5'의 비번역 영역(5'-UTR), 코딩 영역 또는 ORF, 및 3'의 비번역 영역(3'-UTR)의 세 영역이 있습니다. 번역 시작 코돈은 AUG이며 번역 중지 코돈은 3개이며 UAA, UGA, UAG 중 하나입니다. mRNA의 5' 말단에는 5'-5' 결합을 통해 첫 번째 염기에 결합된 캡 (7-메틸 GTP)이 있습니다. 5'-UTR 및 3'-UTR은 비 코딩 엑손 또는 부분적으로 코딩된 엑손의 비 코딩 부분으로 구성되지만 ORF는 코딩 엑손으로 구성됩니다. 일반적으로 3' 말의 마지막 엑손이 가장 길어집니다. mRNA의 3'-UTR은 폴리(A) 신호 서열 5'-AAUAAA-3'을 포함하며, 이는 폴리 아데닐화 부위의 1'-3' 뉴클레오티드 상류에 위치합니다. 포유류의 폴리(A) 꼬리의 길이는 약 200bp입니다. 5'-말단 캡과 3'-말단 폴리(A) 꼬리는 번역을 돕고 mRNA 안정성에도 도움이 됩니다. mRNA의 3'-UTR에 다중 폴리(A) 신호 서열이 포함된 경우, mRNA는 다른 폴리 아데닐화를 받고 매우 다른 안정성을 가진 전사체를 생성할 수 있습니다. 또는, 폴리 아데닐화 mRNA는 3'-UTR 길이도 다릅니다. 그들은 동일한 mRNA의 반감기가 크게 다를 수 있는 다른 조직 또는 다른 발달 단계에서 관찰될 수 있습니다. 17 다중 폴리 (A) 신호 서열을 가진 많은 mRNA가 데이터베이스에 보고되었지만 그 모든 것이 보고 된 것은 아닙니다. mRNA의 5'-UTR는 번역의 시작을 통제합니다. 번역 시작과 올바른 AUG 코돈 (번역 시작 코돈)의 식별과 관련된 중요한 배열을 발견자의 마릴린 코작을 따서 코작 배열이라고 합니다.
나열된 원래 코작 서열은 5'-CCRCCAUGG-3'이고 AUG는 번역 시작 코돈이고 R은 푸딩입니다. 그런 다음 코작 배열의 짧지만 매우 효과적인 버전은 5'-ACCAUGG-3'으로 설명되었습니다. 많은 mRNA에는 컨센서스 코작 서열 또는 이의 변이체가 포함되지만, 코작 서열을 전혀 포함하지 않는 mRNA도 많이 있습니다.
mRNA의 50-UTR 및 30-UTR은 단백질 또는 비 단백질 리간드와 상호 작용하여 유전자 발현과 mRNA의 안정성을 조절할 수도 있습니다. 예를 들어, 페리틴 mRNA의 발현은 5'-UTR에 대한 특정 조절 단백질의 결합에 의해 조절되지만, 트랜스페린 수용체 mRNA의 안정성은 3'-UTR에 대한 특정 조절 단백질의 결합에 의해 조절된다. 합니다. 단백질 리간드와는 달리, 박테리아에서는 특정 mRNA가 특정 비 단백질 리간드에 결합하여 유전자 발현을 조절할 수 있습니다. 소분자에 결합하여 유전자 스위치로 작용하는 mRNA의 부분을 리보스위치라고 합니다. 일부 예로는 보효소 B12 결합 리보스위치, 플라빈 모노뉴클레오티드 (FMN) 결합 리보스위치, 티아민 또는 티아민 피로인산 (TPP) 결합 리보스위치가 있습니다. 이들은 모두 관련 mRNA 5'-UTR에 위치합니다.
2.3 RNA의 2차 구조
RNA 결정 구조 분석은 광범위한 염기쌍의 존재를 밝혀내고 복잡한 3 차 구조 모티프를 많이 생성합니다. Leontis와 Westhof는 왓슨 클릭 에지, 후그 스틴 에지 및 설탕 에지 (2'-OH 포함)의 세 가지 에지 중 하나가 RNA 염기 사이의 평면 에지 간 수소 결합 상호 작용입니다. 참여할 것을 제안했습니다. 염기의 약 60%가 표준 왓슨 클릭 염기쌍에 참여하고 있습니다. 원래의 기하학적 명명법과 분류는 최근 Abu Almakarem 등에 의해 재검토되고 있습니다. 그들은 서로 바꿀 수 있는 반복 염기 트리플렛(이 간행물에서 "염기 트리플"이라고 함)을 식별하는 데 도움이 될 것으로 예상되는 분류 체계를 개발했습니다. RNA의 3차원 구조를 보존하면서. 따라서 이 시스템은 RNA의 3차원 구조 예측 및 RNA 서열 진화 분석에 적용할 수 있습니다. Leontis와 Westhof는 염기가 상호 작용할 수 있는 공간적 배향을 고려하여 염기를 연결하는 적어도 2개의 H-결합을 가진 12개의 기본적인 기하학적 유형을 확인했습니다. 즉, Leontis와 Westhof는 12개의 염기쌍 패밀리를 정의했습니다. 이러한 12개의 염기쌍 패밀리 조합 열거를 사용하여 Abu Almakarem et al. 대표적인 원자분해능의 RNA 3차원 구조를 검색하면 예측된 108개의 베이스 트리플 패밀리 중 68개의 예가 밝혀졌습니다. 또한 모델을 구축한 결과, 나머지 40개 패밀리의 일부는 입체적인 이유로 형성될 수 없는 것일 수 있음을 시사하였습니다.
'생물학' 카테고리의 다른 글
| 시퀀싱(Sequencing) (0) | 2024.03.19 |
|---|---|
| 게놈(genome)의 구성 (0) | 2024.03.16 |
| 세포의 배양(Culture) (0) | 2024.03.15 |
| 원핵세포와 진핵세포 (0) | 2024.03.13 |
| 유전자와 게놈의 기초 (1) | 2024.03.13 |




